Nature Communications I 通过化学机械不稳定性对超灵的敏阈值响应进行编程
通过化学机械不稳定性对超灵的敏阈值响应进行编程
超灵敏阈值响应在生化系统中无处不在,是产生细胞调节紧急行为的重要组成部分。分子滴定是在天然分子网络中发现的一种典型的超灵敏机制。当输入分子被抑制剂隔离或缓冲在非活性复合物中时,就会发生这种情况,然后在所有抑制剂被消耗后出现超灵敏的阈值响应。分子滴定的简单性使其对于将超灵敏度编码为合成构建块具有吸引力。然而,独立控制触发点和阈值响应的灵敏度是有限的,因为它们受抑制剂的浓度和亲和力的影响。因此以可控的方式在合成分子结构中实现超灵敏度是具有挑战性的。2021年8月,Young-Joo Kim, Junho Park, Jae Young Lee, Do-Nyun Kim的研究团队通过使用Park NX10原子力显微镜,在《Nature》杂志上发表了“Programming ultrasensitive threshold response through chemomechanical instability” (https://doi.org/10.1038/s41467-021-25406-9) 的研究文章。他们提出了一种受米歇尔不稳定性(Michell’s instability )启发,通过合理设计 DNA 环的横截面形状和机械性能来控制这种超灵敏阈值重构的临界点和灵敏度。
实验采用了环结构的基于屈曲的结构分叉,称为米歇尔不稳定(图 1a)。环形结构是将一根直杆的两端进行一定的扭转连接而成。两端均受拓扑约束以防止其自由旋转。只要施加的扭曲 (θ) 低于临界值 (θcr ) ,系统就会保持平面环构象。如环的有限元 (FE) 分析结果所示(图 1b ),施加的扭曲纯粹作为扭转应变能存储在结构中,导致零扭曲(W [R]) 和恒定的面内弯曲应变能。然而,当它超过θcr 时,由于米歇尔的不稳定性,环结构突然变得超螺旋并具有非零扭动,其中存储的扭转应变能急剧转变为面外弯曲能。因此,环能够缓冲一定量的扭转应变能,从而在不同的扭转角下实现从圆形到超螺旋的类似开关的阈值重构。为了在分子水平上研究米歇尔不稳定,研究使用 DNA 折纸方法构建了环结构(图 1c)。通过设计将长病毒单链折叠成环的短单链 DNA 序列,可以轻松地对各种横截面和曲率进行编程。DNA嵌入剂被用作化学机械刺激来诱导环的扭转。由于相邻的 DNA 螺旋是交联的,由它们的结合引起的 dsDNA 的几何扰动会在环结构中引起扭转应力,Z终在达到临界值时导致正(即左旋)超螺旋(图 1d)。因此,由于化学机械驱动的米歇尔不稳定性,DNA 折纸环表现出急剧的构象变化。
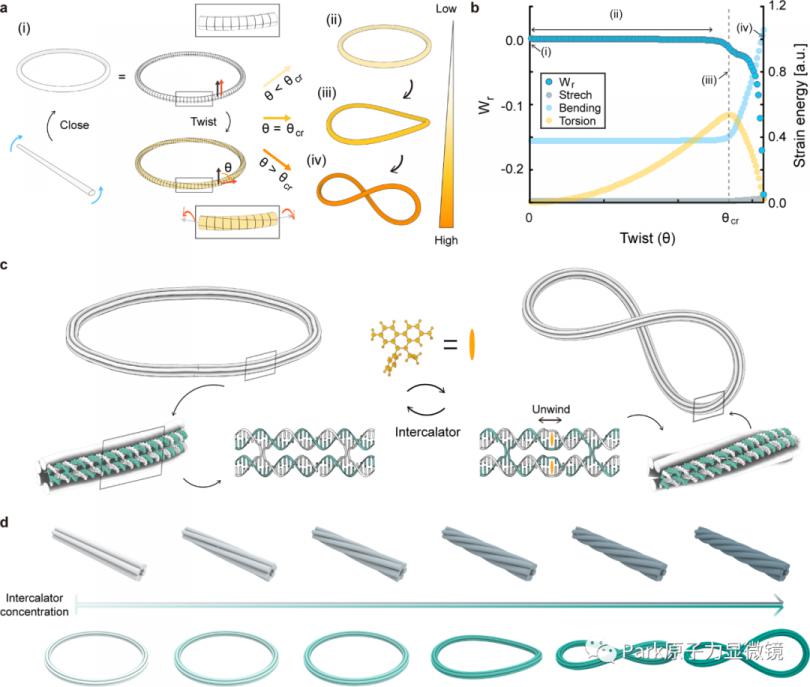
图 1:DNA 环的化学机械不稳定性
为了证明这一点,实验构建了具有和不具有拓扑约束的六螺旋束(6HB)和十螺旋束(10HB)DNA折纸环(图2a)。原子力显微镜 (AFM) 图像用来分析这些环结构的构象变化,以响应模型系统中系统变化的 EtBr 浓度,因为 EtBr 是一种单嵌入剂,在所采用的三种嵌入剂中诱导 DNA 的解旋Z高在这个研究中。在此研究的浓度范围内,EtBr 对 DNA 纳米结构的结合密度为每个 BP 约 0-0.2 个分子。测量了折叠良好的单体中非圆形(卷曲或扭曲)结构(RNC)的比率(图 2b)。对于开环,RNC 随 EtBr 浓度逐渐增加。由于两端可以自由旋转,随着 EtBr 浓度的增加,它们变成左旋螺旋线圈,扭曲率逐渐增加(图 2c)。10HB 闭环的 EtBr 临界浓度将高于 6HB 闭环的临界浓度。该结果表明所提出的控制阈值响应的方法具有很大的灵活性。能够通过 DNA 束环的结构设计来定制屈曲转变的临界浓度,同时保持超敏度水平,这在结构 DNA 纳米技术中已经很好地建立。
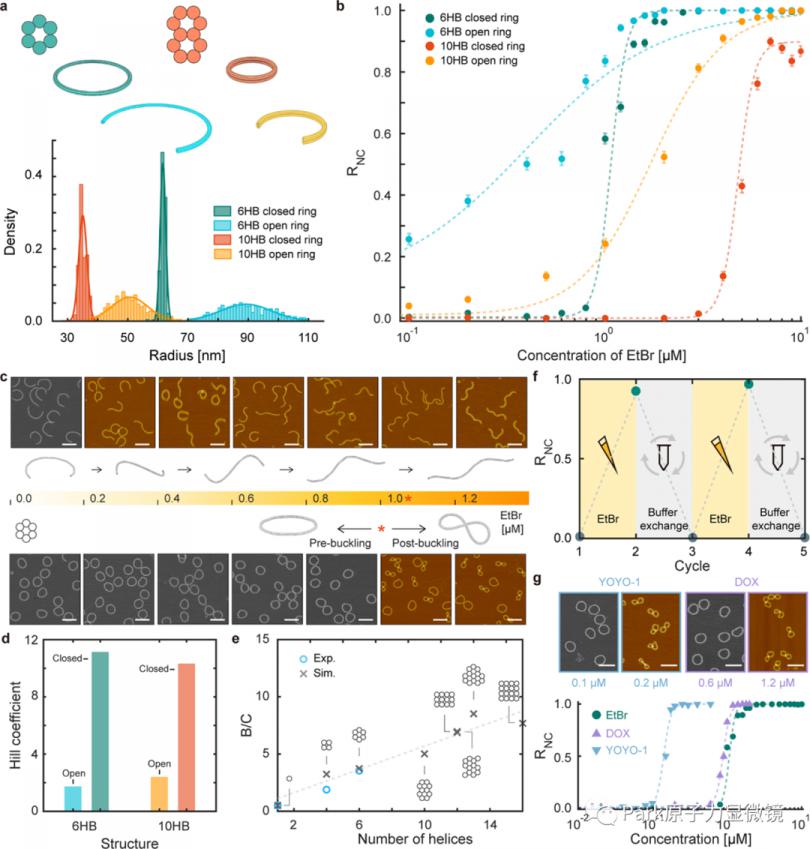
图 2:开放和封闭 DNA 环的重新配置
DNA 折纸环重构的驱动力是由嵌入剂诱导的 DNA 双链体几何扰动引起的化学机械(扭转)应力。可以通过将称为间隙的短单链区域引入结构中来系统地放松 DNA 束中的机械应力。由于重构的敏感性受嵌入剂引起的扭转应力水平控制, 通过使用环的横截面设计和间隙或工程缺陷的灵敏度来独立调节化学机械刺激的临界浓度。
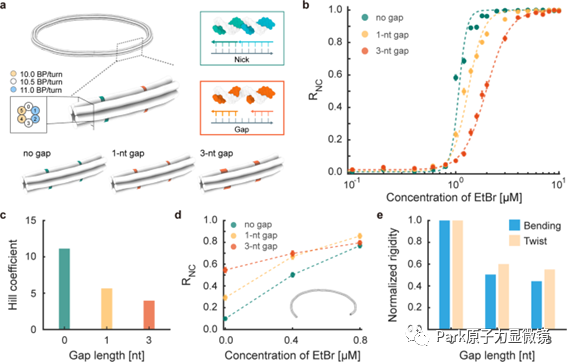
图 3:通过间隙应力松弛来调节灵敏度
基于米歇尔不稳定性的超灵敏阈值响应时,形成封闭结构的拓扑约束和环中结构形状的均匀性都很重要。使用相同的横截面形状和 6HB 闭合环的束长度设计了一个 6HB 闭合三角形。三角形有直边和弯曲的顶点,因此,与环相反,变形能量集中在顶点附近,在那里它在整个结构中分布相当均匀。6HB 闭合三角形在 EtBr 结合后显示出类似于开环的逐渐重构,但没有阈值(图 4a )。即使在低浓度的 EtBr 下,在没有任何诱导扭转应变能缓冲的情况下,也会在顶点处出现局部卷曲模式(图 4b)。闭环和三角形结构的有限元分析也捕捉到了这些行为。它们的三维形状和应变能是通过改变 dsDNA 的螺旋度来计算的,以考虑 EtBr 结合的影响。均方根位移(RMSD)曲线显示两种结构在螺旋度的构象变化方面存在显着差异(图4c)。闭合环在没有显着构象变化的平面中保持其圆形形状,并且随着整个结构的扭转能增加,其应变能几乎均匀地增加(图 4d)。相比之下,闭合三角形通过螺旋度的变化逐渐重新配置,局部卷曲发生在其顶点附近,那里集中了更高的应变能。三角形结构不能保持平面配置,在顶点和边缘处引起不同的变形能,同时仍然满足拓扑约束下的几何兼容性。因此,必须设计一种形状均匀的结构,以便化学机械诱导的变形能可以均匀地分布在整个结构中,以实现超灵敏的阈值响应。
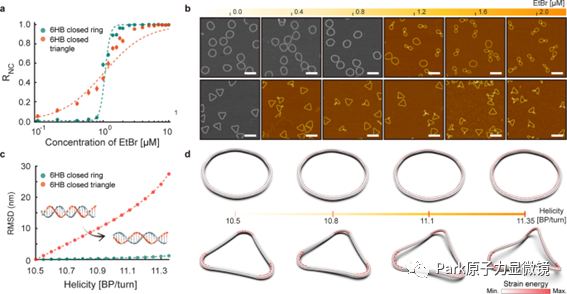
图 4:环和三角形之间重构响应的差异
实验表明,在没有抑制剂分子的情况下,可以通过化学机械不稳定性将超灵敏阈值响应编程到合成分子结构中。由于米歇尔不稳定性,拓扑约束的分子环能够缓冲化学机械诱导的应力而不会发生构象变化。虽然米歇尔不稳定性在理论上被用来解释 dsDNA 环的超螺旋,但在本文中,我们以高度可编程的方式将其应用于结构化 DNA 组装。通过控制给定嵌入剂的环的全局和局部机械性能,可以轻松调节触发点和重新配置的灵敏度。
原子力显微镜成像
本实验AFM 图像由 NX10机台(Park Systems,www.parksystems.cn)利用 SmartScan 软件中的非接触模式拍摄。在测量中使用弹簧常数为 42 N/m 的 PPP-NCHR 探针。每个图像的样本面积为 5 μm × 5 μm,分辨率为 1024 × 1024 像素。通常,每个案例的一个样品获得 5 到 8 个 AFM 图像,以获得统计上足够数量的单体颗粒(至少 300 个颗粒)。使用 XEI程序 (Park Systems) 将图像按线性和二次顺序展平。从 AFM 图像中提取 DNA 折纸结构的单体粒子图像,并手动完成单个粒子的分类。
全部评论(0条)
推荐阅读
-

- Nature Communications I 有机聚合物半导体的电子和纳米机械特性的动态自稳定
- 在环境条件下使用原子力显微镜(AFM)在 C16-IDTBT 薄膜上的350 nm×350 nm区域进行高度及相位图像扫描。
-
- Nature Communications I 2022年Park AFM奖学金项目公布第一位获奖人
- 设计和开发新型性能优异的自粘附导电聚合物材料至关重要。Park NX10 AFM则在此项科研中起到了举足轻重的作用。
-

- 香港中文大学张立团队《Nature Communications》:基于屈曲不稳定性编码的非均质磁化实现软材料结构动态形貌的调控
- 通过利用各式屈曲变形产生的不同微流体行为(如定向流体、混合流体、涡流),该研究结合高精度3D打印技术
-

- Nature Communications:用于远场高分辨成像的新型柔性声学超表面功能器件
- 声学超表面结构通常是刚性而固定的,厚度在毫米以上,甚至与波长相当。并且,这些超表面的工作频率通常在较低的频率,高频高性能应用受限。尽管高精度三维打印技术的快速发展,使得加工更小的超表面构件成为可能。
-

- Nature Communications:用于远场高分辨成像的新型柔性声学超表面功能器件
- 声学超表面结构通常是刚性而固定的,厚度在毫米以上,甚至与波长相当。并且,这些超表面的工作频率通常在较低的频率,高频高性能应用受限。尽管高精度三维打印技术的快速发展,使得加工更小的超表面构件成为可能。
-

- 应用分享|通过DESI-MS对肾上腺进行超高分辨率成像
- 不同质谱仪比较数据表明,MRT质谱仪提供的>200,000 FWHM质量分辨率能够对分子质量非常相似的化合物进行质谱分离,而常规QToF无法解析。
-
- Nature Communications | 亚5nm针尖支撑半悬浮态等离激元超表面的超快纳米光机操纵研究
- 通过低成本大面积的纳米阵列结构制造工艺成功设计并制备了一种亚5nm针尖支撑的超高均一度半悬浮态纳米光机械超表面,并巧妙地实现了光能到机械能的低损耗传输
-
- Nature Communications | 可重复利用的高效圆偏振发光软光子晶体膜
- 国家纳米科学中心段鹏飞研究员团队设计了一种具有可循环利用的手性液晶织构印迹的软光子晶体(SPC)薄膜
-

- 北航文力课题组《Nature Communications》:基于超精密3D打印柔性传感的软体机器人“非接触式”交互示教
- 操作人员通过裸手不仅能够实现对具有大量自由度的软体机器人的非接触控制,而且可以完成各类复杂的操作。
-

- 【安徽大学李亮教授课题组】化学气相沉积生长具有各向异性光热电超快响应的二维晶体PdSe2技术分享
- 可实现在零偏压下探测超宽范围波长甚至是任意波长,该探测器具有超宽范围响应、空气稳定,偏振光响应, 响应度高等优势。
-

- Nature Communications | 一种新型基于自移波性质的二维钙钛矿及其用于PET医学成像
- 近年来,X/γ射线的探测在医学成像、剂量测量、辐射防护等领域已经取得广泛应用。铅卤钙钛矿作为一种新兴的闪烁材料受到了广泛关注。然而,其激子发光的斯托克斯位移较小,且存在严重的自吸收问题,大量激发光子无法被探测到,制约了该材料的探测性能和应用。为解决这一难题,研究人员进行了深入研究,取得了令人振奋的
-

- Nature Communications | 一种新型基于自移波性质的二维钙钛矿及其用于PET医学成像
- 近年来,X/γ射线的探测在医学成像、剂量测量、辐射防护等领域已经取得广泛应用。铅卤钙钛矿作为一种新兴的闪烁材料受到了广泛关注。然而,其激子发光的斯托克斯位移较小,且存在严重的自吸收问题,大量激发光子无法被探测到,制约了该材料的探测性能和应用。为解决这一难题,研究人员进行了深入研究,取得了令人振奋的
-

- Nature Communications | 荧光探针新突破!储军课题组开发出新型高性能基因编码的cAMP荧光探针
- 利用双光子显微镜和光纤记录仪,在果蝇和小鼠等模式生物内,该探针可实时监测特定动物行为过程中特定神经元的cAMP信号的时空动力学变化。
①本文由仪器网入驻的作者或注册的会员撰写并发布,观点仅代表作者本人,不代表仪器网立场。若内容侵犯到您的合法权益,请及时告诉,我们立即通知作者,并马上删除。
②凡本网注明"来源:仪器网"的所有作品,版权均属于仪器网,转载时须经本网同意,并请注明仪器网(www.yiqi.com)。
③本网转载并注明来源的作品,目的在于传递更多信息,并不代表本网赞同其观点或证实其内容的真实性,不承担此类作品侵权行为的直接责任及连带责任。其他媒体、网站或个人从本网转载时,必须保留本网注明的作品来源,并自负版权等法律责任。
④若本站内容侵犯到您的合法权益,请及时告诉,我们马上修改或删除。邮箱:hezou_yiqi














参与评论
登录后参与评论